Das Genom des Bilharziose-Erregers Schistosoma mansoni ist entschlüsselt. Nach der Sequenzierung in den USA hat ein internationales Forscherteam mit mehr als 50 Wissenschaftlern aus sieben Ländern 11.812 Gene identifiziert. Dabei stießen sie auf sehr ungewöhnliche Sequenzen und listeten zudem 34 Gene auf, die Ansatzpunkte für die Entwicklung neuer Medikamente bieten können. Die Ergebnisse des vierjährigen Projekts wurden jetzt in der Fachzeitschrift „Nature“ veröffentlicht.
Die tropische Krankheit Bilharziose (Schistosomiasis) wird durch im Wasser lebende parasitäre Saugwürmer ausgelöst, die durch die Haut in den Menschen eindringen. Weltweit erkranken daran rund 210 Millionen Menschen; in entlegenen Regionen kann die Krankheit ohne Behandlung tödlich verlaufen. Der Wurm Schistosoma mansoni setzt sich für bis zu 25 Jahre in menschlichen Blutgefäßen fest und legt Eier in der Darmwand ab. Sie wandern auch in die Leber, wo sie Geschwulste und eine krankhafte Vermehrung von Bindegewebe auslösen können. Zur Behandlung der Krankheit sind bislang nur zwei Medikamente auf dem Markt, die jedoch nicht vor erneuter Infektion schützen und gegen die die Saugwürmer zunehmend resistent werden könnten. Weil auch die Suche nach einem wirksamen Impfstoff bisher erfolglos blieb, wird nach Ansatzpunkten für neue Therapien gesucht.
Nur fünf Prozent Gene
Die mit der Sequenzierung ermittelte reine Abfolge der Basen im Genom zeigt noch nicht, wo sich die Gene, die entscheidenden Träger der Information, befinden. Sie machen bei S. mansoni insgesamt nur weniger als fünf Prozent der DNA-Sequenz aus; zudem sind bei den meisten Genen die kodierenden Abschnitte, sogenannte Exons, von längeren, nicht-kodierenden Sequenzen, den sogenannten Introns, unterbrochen. Daher ist eine Analyse notwendig, um die Gene zu lokalisieren und ihre Exon-Intron-Struktur zu erkennen.
Als eines von drei Gen- Vorhersageprogrammen kam dabei die an der Universität Göttingen entwickelte Software AUGUSTUS zum Einsatz. Dieses Programm beruht auf einem mathematischen Modell und lernt an einer kleinen Beispielmenge von Genen die typische Basenzusammensetzung in verschiedenen funktionellen Bereichen einer für ein Protein kodierenden DNA-Sequenz.
Zahlreiche ungewöhnliche, rekombinierbare Gene
Aufbauend auf diesem Modell wurde die komplette Genomsequenz von S. mansoni mit mehr als 380 Millionen Basen durchsucht und die Gene, Exons und Introns „vorhergesagt“. Die Kombination und teilweise manuelle Korrektur der vorhergesagten Gene führte die Wissenschaftler zur Sequenz und Struktur von mehr als 11.800 Genen, die nun in einer Datenbank anderen Forschern zugänglich sind. Auf Grundlage der vorhergesagten Proteine untersuchte das Wissenschaftlerteam zudem, welche Genfamilien und Stoffwechselwege in dem Saugwurm existieren.
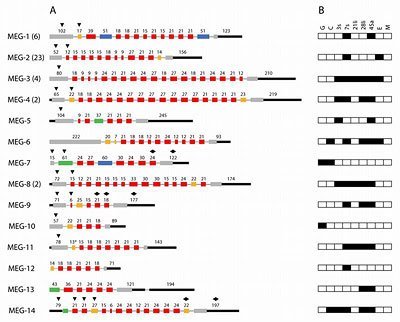
Die Wissenschaftler fanden im Genom von S. mansoni eine Menge von Genen mit ungewöhnlicher Struktur. Diese Gene bestehen aus überwiegend ungewöhnlich kurzen Exons, die jeweils verschieden kombiniert werden können und so dem Wurm die Möglichkeit bieten, mit einem Gen viele verschiedene Varianten eines Proteins zu kodieren. Die von diesen „Mikro-Exon-Genen“ kodierten Proteine haben keine Ähnlichkeit mit Proteinen außerhalb der Gattung Schistosoma und ihre Funktion ist noch unbekannt.
Proteine als Ansatzstellen für mögliche Therapien identifziert
In anderen Fällen konnten die Wissenschaftler durch Vergleich der gefundenen Proteinsequenzen mit bereits bekannten, ähnlichen Proteinen Aussagen über deren wahrscheinliche Funktion machen. So schließen sie aus dem Fehlen von Teilen des Lipidstoffwechsels, dass der Wurm hierfür auf den Wirt angewiesen ist. Zudem fanden sie Neuropeptide, die nicht im Menschen vorkommen. Hier vermuten sie einen möglichen Ansatz für die Entwicklung von Medikamenten, die den Wurm schädigen ohne den Menschen zu beeinträchtigen.
Insgesamt listen die Wissenschaftler in ihrer Publikation 34 Gene in S. mansoni auf, die in ihrer Sequenz Proteinen ähneln, die Ziele von Wirkstoffen in bereits für den Menschen zugelassenen Medikamenten sind. Sie zeigen damit neue mögliche Ansatzpunkte für die pharmazeutische Forschung, um die Krankheit in Zukunft wirksam behandeln und weltweit eindämmen zu können.
(Universität Göttingen, 16.07.2009 – NPO)