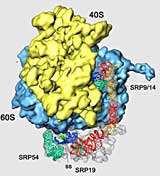
Wissenschaftlern der Humboldt-Universität Berlin ist es mittels Kryo-Elektronenmikroskopie zum ersten Mal gelungen, in der lebenden Zelle einen Bindungsfaktor im aktiven Zustand sichtbar zu machen. „SRP“ spielt eine wesentliche Rolle bei der Herstellung und dem Transport von Proteinen.
1999 erhielt Günter Blobel den Nobelpreis für Medizin für seine Arbeiten zur Proteinsortierung in Zellen. Dabei geht es um ein fundamentales Problem der Zellbiologie: Alle Proteine werden in der Zelle synthetisiert, aber um ihre Wirkung entfalten zu können, müssen viele dieser Moleküle erst einen bestimmten Ort innerhalb oder außerhalb der Zelle erreichen. Im Rahmen seiner Signalhypothese hatte Blobel vorgeschlagen – und später zeigen können -, dass Proteine wie zum Beispiel Antikörper oder Insulin, die außerhalb der Zelle ihre Wirkung entfalten sollen, über so etwas wie einen „Adresszettel“ verfügen. Dieser Adresszettel entspricht einer bestimmten Aminosäureabfolge im Protein – die so genannte Signalsequenz. Er sorgt dafür, dass die entsprechenden Proteine tatsächlich durch eine Membran aus der Zelle hinausgeschleust werden.
Transportvorgang mit Proteinsynthese
Interessanterweise wird dieser Transportvorgang – die Translokation – in Zellen höherer Organismen (so auch beim Menschen) mit der Herstellung des Proteins gekoppelt: Beides findet also gleichzeitig statt. Das bedeutet, dass die ganze Proteinsynthese-Maschine der Zelle, das Ribosom, zu einer entsprechenden Translokationsstelle an der Membran gebracht wird, sobald eine Signalsequenz erkannt wird. Dies wiederum hat den Vorteil, dass Proteine, die aus der Zelle geschleust oder in der Zellmembran verankert werden sollen, schon bei der Fertigstellung sich am richtigen Ort oder auf dem richtigen Weg befinden.
An dieser Stelle gelangt man nun zu dem Schlüssel jenes cleveren Sortierungsprinzips. Dieser Schlüssel ist ein Bindungsfaktor, der den Adresszettel – also die Signalsequenz – erkennt und der daraufhin die Synthese-Maschine an die Membran dirigiert. Ein solcher Bindungsfaktor wurde von Blobel bereits 1971 postuliert und Anfang der achtziger Jahre in seinem Labor auch identifiziert. Der Faktor wurde „Signalerkennungs-Partikel“ oder auch SRP (signal recognition particle) genannt. Er kommt in allen lebenden Zellen vor und besteht wie das Ribosom aus Ribonukleinsäure (RNA) und Proteinen. SRP erkennt tatsächlich Signalsequenzen, bindet daraufhin das Ribosom und dirigiert es mit Hilfe eines membranständigen SRP-Rezeptors zur Membran. Aber SRP kann noch mehr: Sobald es an das Ribosom gebunden hat, wird die Maschine gestoppt, und die Synthese kommt für kurze Zeit zum Erliegen. Erst wenn es an der Membran angelangt ist, wo das neu gebildete Protein – wie oben beschrieben – während seiner Herstellung sofort durch einen Kanal hinausgeschleust werden soll, startet die Synthese aufs Neue. Dabei ist bislang völlig unklar, wie SRP mit der Signalsequenz und dem Ribosom wechselwirkt und wie es insbesondere das Ribosom dazu zwingt, eine Synthesepause einzulegen.
SRP erstmals in Aktion beobachtet
Der Nachwuchsforschergruppe um Roland Beckmann vom Institut für Biochemie der Humboldt-Universität Berlin ist es nun mittels Kryo-Elektronenmikroskopie gelungen, SRP erstmals in Aktion sichtbar zu machen – und zwar an einem gestoppten Ribosom mit Signalsequenz. Zugleich präsentieren sie damit ein räumliches Modell für die molekulare Struktur von SRP in diesem funktionellen Zustand. Hierzu halten wir auf unserer Website unter unten angegebener Adresse eine Abbildung für Sie bereit.
Man kann erkennen, dass SRP ein sehr lang gestreckter Partikel ist und mit der Signalsequenz direkt an jener Stelle am Ribosom, wo das neue Protein hervortritt, wechselwirkt. Gleichzeitig reicht das andere Ende des SRP-Moleküls bis zu einer Stelle, an die normalerweise so genannte Elongationsfaktoren andocken (sie dienen der Herstellung notwendiger Hilfsproteine). Das bedeutet: Aus der Struktur lässt sich ableiten, über welchen Mechanismus SRP die Synthesepause bewirken kann – indem es eben den Elongationsfaktoren einfach den Zugang zur Maschine versperrt. Dabei ist die Form der Interaktion von SRP mit dem Ribosom an dieser Stelle vergleichbar mit jener der Elongationsfaktoren. SRP tut also so, als wäre er selbst ein solcher Faktor – und kann dadurch das Ribosom „übernehmen“.
Darüber hinaus scheint nach Ansicht der Wissenschaftler die Art und Weise der Interaktion von SRP mit dem Ribosom in allen Zellen so zu sein wie hier beobachtet, denn der wichtigste Interaktionspartner ist eine in allen Organismen vorhandene und kaum variable Untereinheit des SRP-Partikels: das SRP54. Es tritt direkt mit der Signalsequenz in Kontakt. „Wir können also einen wichtigen Teil des von Blobel 1971 vorgeschlagenen Prinzips mit molekularen Strukturen und Mechanismen erklären und verstehen nun besser, wie die ersten Schritte der Proteinsortierung – etwa die Signalsequenz-Erkennung – tatsächlich vor sich gehen“, zieht Roland Beckmann ein Fazit seiner Arbeit.
(idw – VolkswagenStiftung, 27.02.2004 – DLO)