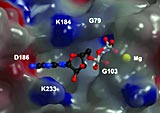
Die erste Struktur einer neuen Proteinklasse (p47) hat ein Wissenschaftler-Team unter Beteiligung von Chemikern der Ruhruniversität Bochum aufgeklärt. Wie die Forscher mit Hilfe der Röntgenkristallographie an Mäusen zeigen konnten, bildet der Organismus diese Proteine als Immunantwort auf den Befall durch Parasiten oder andere Erreger.
Mit der nun publizierten molekularen Charakterisierung und dreidimensionalen Struktur eines dieser Proteine (IIGP1) sind erste Details des Wirkmechanismus erkennbar. Insbesondere Zusammenhänge zwischen der Bindung von Hilfssubstanzen (Co-faktoren) und strukturellen wie funktionellen Änderungen des Proteins werden deutlich. „Die Ergebnisse können in zukünftigen Studien genutzt werden, um weiteren Fragestellungen zur Funktionsweise und zu Wechselwirkungen mit anderen zellulären Partnern gezielt auf den Grund zu gehen“, so Prof. Herrmann.
Protein dient der Abwehr von Erregern
Die neue Klasse anti-parasitärer Proteine wurde vor wenigen Jahren von einer Kölner Arbeitsgruppe identifiziert. Ihre Bildung in der Zelle wird durch das bei Infektionen ausgeschüttete Immunstimulans Interferon angeregt. Mit der Identifizierung und Beschreibung entscheidender Funktionselemente auf molekularer Ebene ist nun der erste Schritt zum Verständnis getan, wie genau diese Proteine in der lebenden Zelle zur Abwehr von Erregern beitragen. „Eine Besonderheit, die erkennbar wurde, ist das Wechselspiel von enzymatischer Aktivität, Co-faktorbindung und dadurch hervorgerufene Strukturveränderungen, die die Proteine anregen, sich selbst zu Aggregaten zusammenzubauen und vermutlich auch spezifische Interaktionen mit noch unbekannten Partnern zur Folge haben“, erläutert Prof. Herrmann. Weitere Untersuchungen haben gezeigt, welche Aminosäuren im einzelnen für die Wechselwirkung, die Co-faktorbindung und -spaltung verantwortlich sind.
Rückschlüsse auf andere Mitglieder der Proteinfamilie
Zum Gelingen der Arbeit haben neben biophysikalischen und strukturellen Methoden auch bioinformatische Techniken und die Nutzung von Genom-Daten beigetragen. „Dadurch ist es uns vor allem gelungen, die hier gewonnenen Erkenntnisse auch für die Erforschung verwandter Proteine einzusetzen oder bestimmte Vorhersagen bezüglich ihrer Funktion und Struktur zu treffen“, so Prof. Herrmann. Diese Ergebnisse bieten konkrete Anhaltspunkte, um in weiteren Studien die molekularen Wirkmechanismen dieser Proteinfamilie und ihre Wechselwirkungen mit zellulären oder parasitären Proteinen aufzuklären. Nicht zuletzt dürfe man sich von diesen und zukünftigen Ergebnissen auch Anwendungsmöglichkeiten und weitere Erkenntnisse für die Medizin erhoffen, so der Forscher.
(idw – Ruhr-Universität Bochum, 11.10.2004 – DLO)