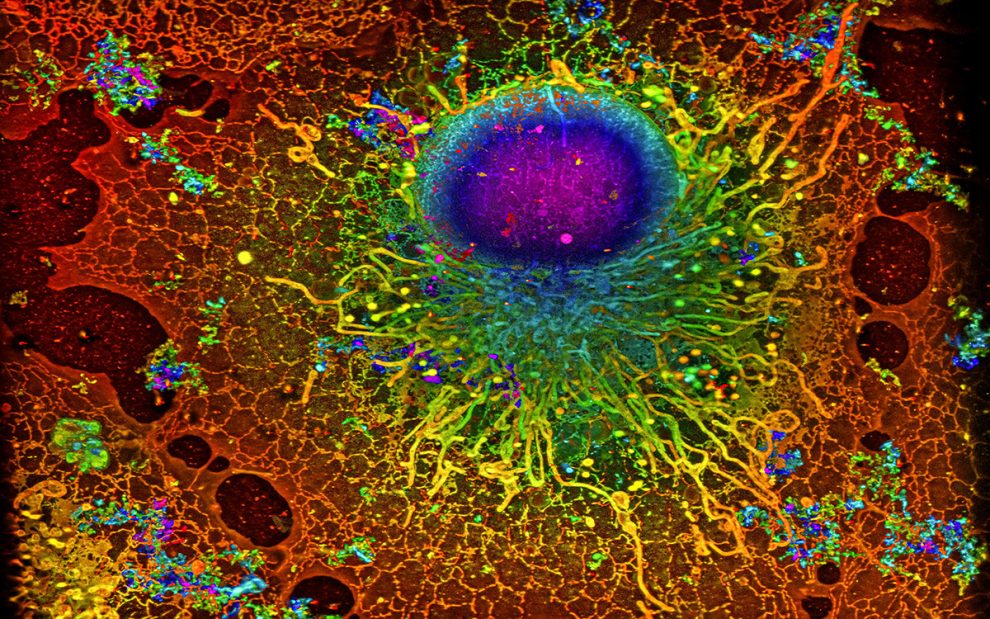
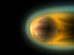
Mehr als nur ein Membranbeutel mit Kern: Wie komplex das Innere einer Zelle aufgebaut ist, verdeutlicht diese hochauflösende Fluoreszenzaufnahme. Sie zeigt eine Bindegewebszelle, deren fluoreszierende Marker erst mithilfe der Einzelmolekül-Lokalisationsmikroskopie aufgenommen wurden, dann wurden die Einzelaufnahmen durch einen lernfähigen Algorithmus zum fertigen Bild zusammengesetzt.
Mit neuen Methoden der superauflösenden Lichtmikroskopie können Wissenschaftler immer tiefer in die kleinsten Strukturen des Lebens hineinblicken. Eine besondere Rolle dabei spielen die verschiedenen Spielarten der Fluoreszenz-Mikroskopie, bei der der Probe molekulare Marker zugesetzt werden, die bei Anregung durch Licht farbig nachleuchten. 2014 gab es für zwei dieser Verfahren den Chemie-Nobelpreis, inzwischen erreichen modernste Fluoreszenz-Mikroskope sogar schon Auflösungen von einem Nanometer.
Eine Variante der superauflösenden Mikroskopie ist die sogenannte Einzelmolekül-Lokalisationsmikroskopie (Single-Molecule Localisation Microscopy, SMLM). Dabei werden pro Aufnahme jeweils nur einige der in der Probe vorhandene Fluoreszenzmarker mit Licht aktiviert – bei jedem Bild sind dies andere. Diese Rohdaten werden dann ausgewertet und zu einem einzigen aussagekräftigen Bild zusammengesetzt.
Lernfähiger Algorithmus als Helfer
Mit der SMLM-Technik lassen sich extrem hohe Auflösungen erreichen, allerdings ist das Verfahren sehr aufwändig. Es benötigt eine große Menge an Bildern, die dann korrekt zusammengesetzt werden müssen. Deshalb haben nun Artur Speiser von der Universität Tübingen und seine Kollegen einen lernfähigen Algorithmus entwickelt, der die Auswertung und Kombination solcher SMLM-Aufnahmen eigenständig übernehmen kann.
Der Algorithmus DECODE (DEep COntext DEpendent) basiert auf Deep Learning: Er nutzt ein künstliches neuronales Netz, das durch Trainingsdaten lernt. Statt mit echten Bildern wird das Netz in diesem Fall jedoch mit synthetischen Daten aus Computersimulationen trainiert, die den realen Aufnahmen sehr ähnlich sind. „Das neuronale Netz, das wir mit Simulationsdaten trainiert haben, ist so in der Lage, fluoreszierende Moleküle auch in echten Bildern zu erkennen und zu lokalisieren“, erklärt Speiser.
Zelldetails in höchster Auflösung
Ein Beispiel für eine von DECODE ausgewertete SMLM-Bildgebung ist hier zu sehen. Sie zeigt eine sogenannte Cos7-Zelle mit allen zellinternen Details. Dabei handelt es sich um in Zellkultur gezüchtete Abkömmlinge von Bindegewebszellen aus der Niere, die in Labors weltweit zur Erforschung und Kultur von Viren und für die genetische Forschung verwendet werden. Zu erkennen ist nicht nur der Zellkern, sondern auch die unzähligen feinen Kanälchen und Strukturen, die das Zellplasma durchziehen.
Der neue Algorithmus kann Fluoreszenzmarker bei höheren Dichten lokalisieren als bislang möglich, so dass weniger Bilder je Probe benötigt werden. Auf diese Weise kann die Bildgebungsgeschwindigkeit ohne nennenswerten Verlust an Auflösung um das bis zu Zehnfache gesteigert werden. Darüber hinaus kann DECODE die Unsicherheiten quantifizieren ‒ das Netzwerk kann also selbst erkennen, wenn es sich in seiner Lokalisierung nicht sicher ist. (Nature Methods, 2021; doi: 10.1038/s41592-021-01236-x)
Quelle: Universität Tübingen