Zahlreiche Krankheiten sind auf falsch gefaltete Proteine, bestehend aus Aminosäuren, zurückzuführen. Forscher haben jetzt mit einer speziellen Spektroskopie diese fehlerhafte Proteinfaltung untersucht. Wie sie in „Nature“ berichten, sind Fehlfaltungen häufiger, wenn die Abfolge der Aminosäuren in den benachbarten Domänen sehr ähnlich ist.
Proteine sind die zentralen molekularen Maschinen in unserem Körper. Um ihre hochspezifischen Funktionen erfüllen zu können, müssen sie eine wohldefinierte dreidimensionale Struktur annehmen. Dieser Prozess der Proteinfaltung kann jedoch fehlschlagen, was zu Funktionsausfällen oder sogar Verklumpungen führt, die neurodegenerative Krankheiten wie Morbus Alzheimer oder Parkinson auslösen können. Forscher der Universitäten Zürich und Cambridge haben jetzt eine spezielle spektroskopische Methode verwendet, die Einzelmolekülfluoreszenzspektroskopie, um herauszufinden, unter welchen Voraussetzungen Fehlfaltung stattfindet.
Muskelprotein Titin als Untersuchungsobjekt
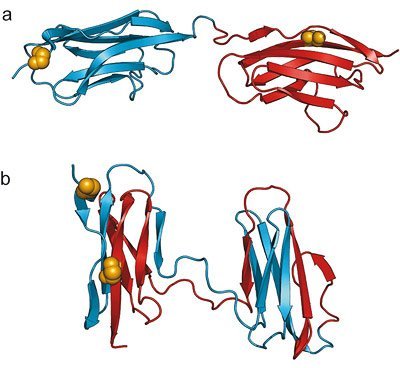
Die Gruppe um Professor Benjamin Schuler von der Universität Zürich untersuchte dafür Teile des größten Proteins unseres Körpers, des Titins. Dieses trägt in den Muskeln zur Stabilität und Elastizität der Muskelfasern bei. Man geht davon aus, dass sich bei starker Belastung des Muskels einzelne Teile des Titins, die Domänen, entfalten, um eine Beschädigung des Muskelgewebes zu verhindern. Wenn sich der Muskel wieder entspannt, besteht die Gefahr, dass sich diese entfalteten Domänen falsch zusammenlagern. Diese Gefahr besteht in ähnlicher Form auch für andere Proteine, die aus mehreren Domänen bestehen.
Für ihre Studie brachten die Forschenden kleine Farbstoffmoleküle als Sonden im Protein an. „Mit unserer laserspektroskopischen Methode konnten mittels Energietransfer zwischen den Sonden Abstände auf molekularer Skala bestimmt werden, also im Bereich einiger Millionstel Millimeter“, erklärt Schuler. So ließen sich die Strukturen richtig und falsch gefalteter Proteine unterscheiden und damit der Anteil an Fehlfaltung bestimmen.
Mehr Fehler bei ähnlichen Aminosäuren
„Die Untersuchung verschiedener Domänen des Titins in unseren Experimenten hat gezeigt, dass die Wahrscheinlichkeit der Fehlfaltung steigt, wenn benachbarte Domänen sich in der Abfolge ihrer Aminosäuren sehr ähnlich sind“, so Schuler. Dies ist anscheinend der Grund dafür, dass in Proteinen die benachbarten Domänen eine geringe Ähnlichkeit haben. „Offenbar handelt es sich dabei um eine wichtige evolutionäre Strategie, um die Fehlfaltung von Proteinen zu vermeiden und so ihre maximale Funktionalität zu gewährleisten“, so Schuler.
(Universität Zürich, 01.06.2011 – NPO)