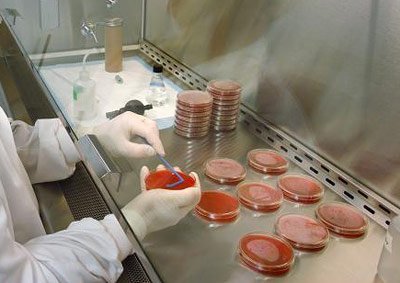
Wenn man früher Bakterien aus einem speziellen Lebensraum untersuchen wollte, musste man versuchen, diese zunächst im Labor zu kultivieren. Anschließend konnte man die gewachsenen Bakterienkulturen genauer analysieren und identifizieren. Mit der Entwicklung molekulargenetischer Methoden sind diese aufwändigen Verfahren nicht mehr nötig. Sie werden heute durch sogenannte metagenomische Methoden ersetzt.
Dabei wird Bakterien-DNA direkt aus einer Probe gewonnen, die ihrem normalen Lebensraum entnommen wurde. Diese Probe wird sequenziert. Aus dem resultierenden DNA-Muster können die Forscher ablesen, welche Bakterien in der Probe enthalten sind. Solche metagenomischen Verfahren sind nicht nur weniger aufwändig als frühere Methoden, sie lassen sich auch ohne weiteres für jede beliebige Probe anwenden. Zudem können mit dieser Methode weitaus mehr Bakterien-Spezies identifiziert werden als durch Kultivierung. Viele Bakterien sind sogar erst anhand ihrer DNA-Sequenzen entdeckt worden.
Ein Gen dient als Erkennungscode
Das Gen der Wahl für metagenomische Studien ist das sogenannte 16S rRNA-Gen. Dieses Gen kommt in allen Organismen und damit auch allen Bakterien vor. In seiner DNA enthält es einige konservierte Regionen, die bei allen Lebewesen gleich sind. An ihnen lässt sich das Gen erkennen. Zusätzlich finden sich im 16S rRNA-Gen aber auch variable DNA-Abfolgen, die im Laufe der Evolution stark verändert wurden. Sie ermöglichen es, die Bakterien zu identifizieren, die dieses Gen tragen.
Im Allgemeinen vermehren die Wissenschaftler zunächst einen Teil des 16S rRNA-Gens aus einer Probe. Anschließend wird entweder der solcherart vermehrte Pool von Sequenzen kloniert und die Sequenz einzelner Klone bestimmt, oder es werden modernste Sequenziergeräte (next-generation sequencing platforms) für die Sequenzierung benutzt. Die so aus einer Probe gewonnenen 16S rRNA-Sequenzen können dann mit einer Datenbank verglichen werden, um die Zugehörigkeit der Bakterien und ihre Häufigkeit in der Probe zu bestimmen.
Mark Stoneking, MPI für evolutionäre Anthrolopologie / Redaktion scinexx
Stand: 28.10.2011